
The 3′ mRNA End Processing Is Critical for Termination of Transcription
 المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
 المصدر:
LEWIN’S GENES XII
المصدر:
LEWIN’S GENES XII
 الجزء والصفحة:
الجزء والصفحة:
 17-5-2021
17-5-2021
 2922
2922
The 3′ mRNA End Processing Is Critical for Termination of Transcription
KEY CONCEPTS
- Transcription can be ended in a number of different ways based on the type of RNA polymerase involved.
- mRNA 3′ end formation signals termination of Pol II transcription.
Information about the termination reaction for eukaryotic RNA polymerases is less detailed than our knowledge of initiation. The 3′ ends of RNAs can be generated in two ways. Some RNA polymerases terminate transcription at a defined terminator sequence in DNA, as shown in FIGURE 1. RNA polymerase III appears to use this strategy by having a discrete oligo(dT) sequence to signal the release of Pol III for transcription termination.
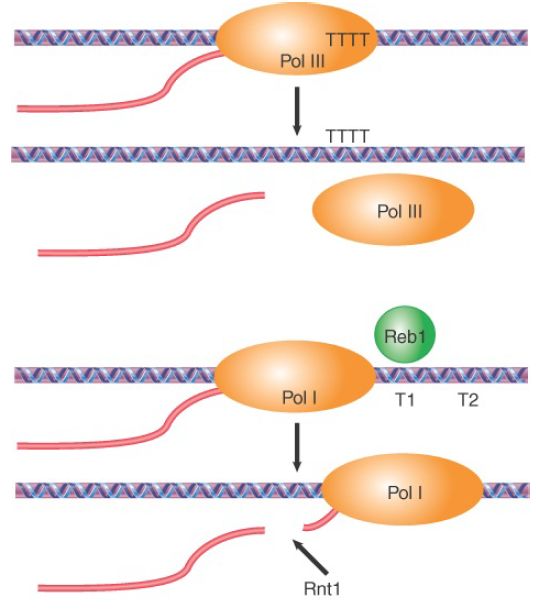
FIGURE 19.31 Transcription by Pol III and Pol I uses specific terminators to end transcription.
For RNA polymerase I, the sole product of transcription is a large precursor that contains the sequences of the major rRNA. Termination occurs at two discrete sites (T1 and T2) downstream of the mature 3′ end. These terminators are recognized by a specific DNA-binding Reb1 in yeast or TTF1 in mice. Pol I termination is also associated with a cleavage event mediated by the endonuclease Rnt1p, which cleaves the nascent RNA about 15 to 50 bases downstream from the 3′ end of processed 28S rRNA . In this regard, Pol I termination is mechanistically related to Pol II termination in that both processes may involve an RNA cleavage event.
In contrast to Pol I and Pol III termination, RNA polymerase II usually does not show discrete termination, but continues to transcribe about 1.5 kb past the site corresponding to the 3′ end. The cleavage event at the polyadenylation site provides a trigger for termination by RNA polymerase II, as shown in FIGURE 2.
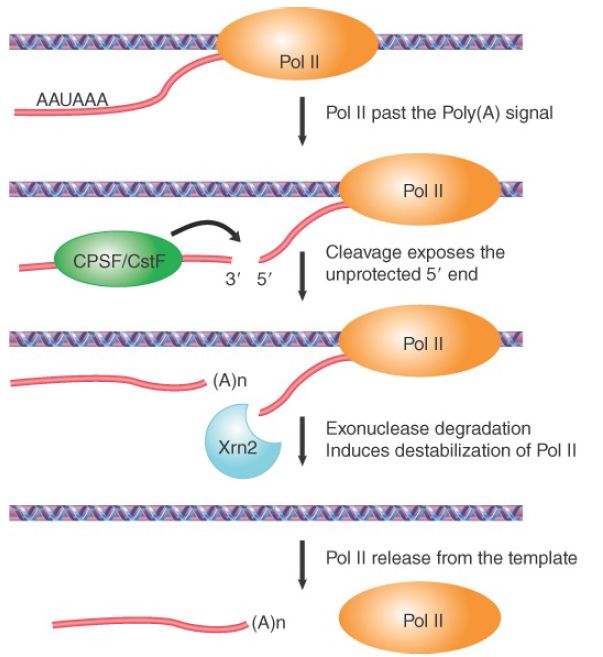
FIGURE 2. 3′ end formation of Pol II transcripts facilitates transcriptional termination.
Two models have been proposed for Pol II termination. The allosteric model suggests that RNA cleavage at the polyadenylation site may trigger some conformational changes in both the Pol II complex and local chromatin structure. This may be induced by factor exchanges during the polyadenylation reaction, resulting in Pol II pausing and then release from template DNA.
An alternative model known as the torpedo model proposes that a specific exonuclease binds to the 5′ end of the RNA that is continuing to be transcribed after cleavage. It degrades the RNA faster than it is synthesized, so that it catches up with RNA polymerase. It then interacts with ancillary proteins that are bound to the carboxy-terminal domain of the polymerase; this interaction triggers the release of RNA polymerase from DNA, causing transcription to terminate. This model explains why the termination sites for RNA polymerase II are not well defined, but may occur at varying locations within a long region downstream of the site corresponding to the 3′ end of the RNA. The major experimental evidence for the torpedo model is the role of the nuclear 5′–3′ exonuclease Rat1 in yeast or Xrn2 in mammals. Deletion of the gene frequently causes readthrough transcription to the next gene.
However, in some experimental systems, mutation of the AAUAAA signal to impair cleavage at the natural polyadenylation site does not necessarily trigger the release of the transcribing Pol II and cause transcriptional readthrough. This evidence, coupled with some local changes in chromatin structure, thus favors the allosteric model.
It has become apparent that the allosteric and torpedo models are not necessarily mutually exclusive; both may reflect some critical aspects associated with Pol II transcriptional termination. By either or both mechanisms, it is clear that transcriptional termination by Pol II is tightly coupled with the 3′ end formation for most mRNAs in eukaryotic cells.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة


